本站讯 近日,中国海洋大学医药学院、青岛海洋科技中心海洋药物与生物制品实验室、海洋药物教育部重点实验室于日磊教授联合中国科学院生物物理研究所赵岩研究员,在药学领域知名学术期刊Acta Pharmaceutica Sinica B(药学学报)在线发表题为“Deep Learning-Driven Discovery and Mechanism of Action Study of a Minimalist Conopeptide Targeting α7 Nicotinic Acetylcholine Receptor”(深度学习驱动的靶向α7烟碱型乙酰胆碱受体的极简芋螺肽发现及其作用机制研究)的研究文章。
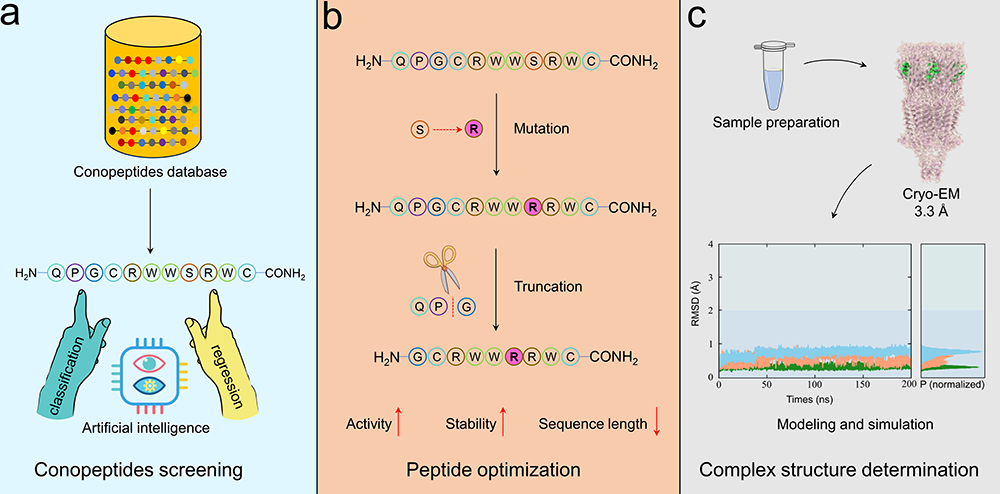
图1 基于人工智能的多肽发现与活性优化平台
芋螺能产生多种生物活性肽,包括富含二硫键的芋螺毒素和不含二硫键的芋螺多肽,为药物研发及神经化学工具的开发提供了宝贵资源。尽管现代组学方法已显著提升了芋螺多肽的发现效率,但其相应的药理学研究进展仍相对滞后。目前,对功能特异性芋螺肽的筛选主要依赖复杂的电生理实验进行随机筛选。近年来,基于组学与噬菌体展示等高通量筛选技术的引入,显著提高了生物活性肽的发掘效率。然而,建立快速、高通量且具备功能导向的筛选策略,对于芋螺多肽的功能解析与高效挖掘仍具有重要的科学意义和应用价值。
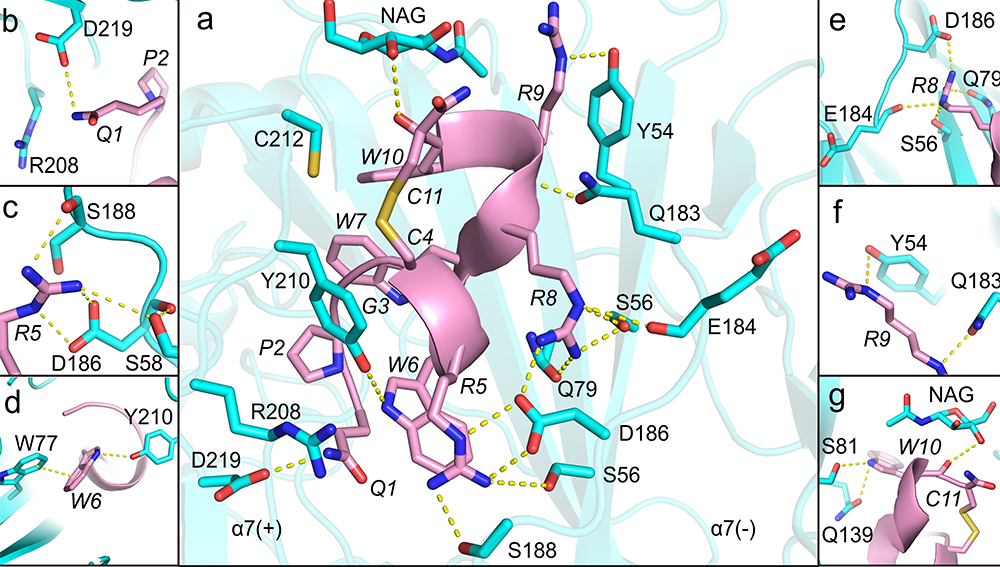
图2 优化多肽分子α7 nAChR与[S8R]SS1的结合模式图
研究团队提出一种结合深度学习、结构生物学、计算建模和药物化学的创新策略,成功实现了靶向α7 nAChR(一种关键的离子通道受体,与多种神经退行性疾病密切相关)的芋螺多肽发现与优化进程。在此项研究中,团队构建了基于ESM-2蛋白语言模型的深度学习筛选框架,从689种不含二硫键的芋螺多肽中高效筛选出活性候选分子SS1。在此基础上,通过构效关系研究对其进一步优化,获得了活性显著提升的[S8R]SS1,该简化多肽具备纳摩尔级的抑制活性,并展现出优良的选择性与稳定性。为深入探索其作用机制,联合研究团队利用冷冻电子显微镜技术,解析了α7 nAChR与[S8R]SS1的复合物三维结构,并结合分子动力学模拟揭示其独特的结合模式和作用机制。本研究不仅为α7 nAChR靶向海洋多肽的开发提供了全新的方法学支撑,也为药物发现领域开辟了更为高效的路径。尤为重要的是,该工作展示了在数据匮乏条件下,如何借助深度学习实现功能导向的天然多肽筛选,并利用计算建模和分子模拟有效弥补了冷冻电镜在解析高柔性多肽-受体复合物结构时的局限,从而显著推动天然多肽药物的研发进程。

研究团队合影(第一排左四为于日磊教授)
中国海洋大学为上述研究的第一完成单位,医药学院于日磊教授和中国科学院生物物理研究所赵岩教授为共同通讯作者,中国海洋大学博士研究生张景会、尹正基、葛成和中国科学院生物物理研究所博士研究生李悦为共同第一作者。该研究得到了国家自然科学基金等项目资助。
文章链接:https://doi.org/10.1016/j.apsb.2025.12.035
编辑:赵奚赟
责任编辑:刘莅




































