本站讯 近日,中国海洋大学海洋生物遗传学与育种教育部重点实验室包振民院士和王师教授团队在国际生物信息数据库领域知名期刊Nucleic Acids Research(《核酸研究》)发表了题为“Ocean-M: an integrated global-scale multi-omics database for marine microbial diversity, function and ecological interactions”(面向全球格局的海洋微生物综合多组学资源数据库—Ocean-M)的论文,正式发布了面向全球格局的海洋微生物综合多组学资源数据库—Ocean-M。
海洋微生物是海洋物质循环与能量流动的核心,支撑全球生态系统的平衡与健康。它们在极端环境中的适应能力为揭示生命进化提供了重要视角,同时其所蕴藏的丰富生物活性物质,在海洋资源开发、环境污染治理和生物技术等领域具有广泛的应用潜力。随着海洋微生物多维度组学数据的快速增长,全球海洋微生物学研究正面临前所未有的数据整合与深度分析挑战。现有数据库多以数据资源存储和分析工具为主,缺乏对多源异构数据的系统挖掘与深度解析,如何实现“复杂、高维、海量”的海洋微生物数据的系统整合,实现从“单一物种”到“多维生态位”的全面深度解析仍是亟待突破的难题。为应对这一挑战,研究团队开发了Ocean-M平台,构建了全球海洋微生物多组学综合资源数据库Ocean-M,打破了传统数据库的局限,形成了“组学资源—生态互作—功能挖掘”一体化深度研究平台。
Ocean-M数据库整合了全球多生境、多尺度的海洋微生物多组学数据,成为海洋微生物研究领域最全面的开放获取数据库。平台汇集了来自全球不同海洋生态系统的57,000余个微生物数据,涵盖宏基因组、宏转录组、宏蛋白质组、宏代谢组等多个层次,样本涵盖了从极地到赤道、从表层到深海的完整生态梯度。通过统一的数据处理和标准化标识符,Ocean-M整合了规模最大的宏基因组(MAGs)数据,包含超过50,000个代表性微生物基因组数据,支持全球范围内微生物群落结构、功能特征及其空间分布的可视化展示。Ocean-M构建了基于全息组学的生态网络分析框架,整合了微生物群落网络、宿主-微生物组互作及环境DNA数据集,能精确描述查询微生物群落结构、功能特征及其生态位相互关系,实现了生态互作的全维度分析,为生态学研究提供了全新的视角和工具。同时,搭建了“基因组-基因功能-生态偏好”三位一体的系统分析框架,通过大规模的功能基因挖掘,建立了包含百万级功能基因集合,为微生物功能基因的挖掘和生态系统功能解析提供了强有力的支持。此外,团队在前期开发的微生物精准鉴定方法2bRAD-M的基础上,打造了超5万海洋微生物组装基因组(MAGs)的特异性2b-RAD标签库,可鉴定物种达95%以上,为复杂海洋环境中微生物的精准识别和分类提供解决方法。Ocean-M不仅为海洋微生物学、生态学等基础科学研究提供了强大的数据支持,还为海洋生物资源开发和生物技术创新等领域提供了宝贵的数据资源。
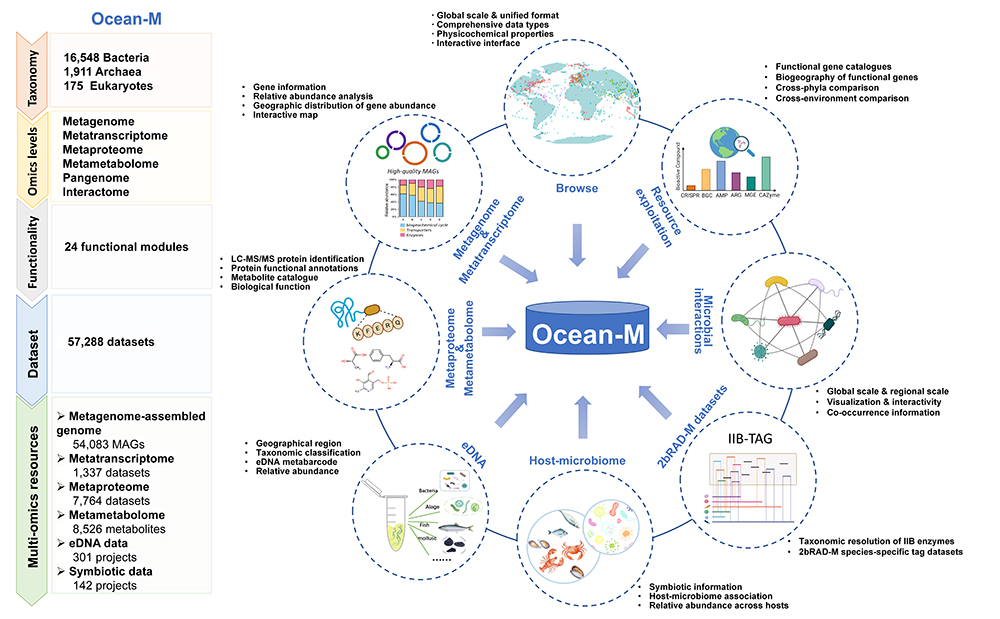
Ocean-M数据库架构和功能模块概览
海洋生物遗传学与育种教育部重点实验室、方宗熙海洋生物进化与发育研究中心的王师教授、包振民院士和张玲玲教授为论文的共同通讯作者,吕佳副教授、硕士生马帅康、马岑博士和刘福云博士为论文共同第一作者。该研究工作获得中国海洋大学张玉忠教授团队、哈佛医学院孙政博士、中国科学院微生物研究所王军研究员、青岛华大基因研究院范广益博士、香港大学黄适教授、北京百迈客生物科技有限公司、中国海洋大学高性能生物超算中心等的支持与协助,以及国家重点研发计划、崂山实验室科技创新项目、国家自然科学基金项目资助。
文:王志刚
文章链接:https://doi.org/10.1093/nar/gkaf1098
编辑:赵奚赟
责任编辑:刘莅




































