本站讯 11月12日,中国海洋大学“海洋生物遗传学与育种”教育部重点实验室包振民院士和王师教授团队在国际生物信息数据库领域顶级期刊Nucleic Acids Research在线发表了国际上迄今最大规模的软体动物功能和进化基因组学综合数据库“MolluscDB 2.0: a comprehensive functional and evolutionary genomics database for over 1400 molluscan species”(MolluscDB 2.0:面向超过1400个软体动物种类的功能和进化基因组学综合数据库)。
软体动物(Mollusca)是动物界第二大门类,也是最大的海洋动物门类。软体动物起源于5亿年前早寒武纪,现存种类超过10万种,是进化上最成功的无脊椎动物群体之一。此外,许多软体动物也是重要水产经济物种,其占世界水产总产量高达27%。随着软体动物基因组学迅速发展,取得的重要科学发现层出不穷,极大地提升了对动物起源和适应性演化的认知深度。联合国内外优势机构,本研究团队于2021年发起了国际万种软体动物基因组计划(M10K project),并构建了国际首个软体动物综合基因组数据库MolluscDB(http://mgbase.qnlm.ac)。该数据库整合了约1000份软体动物基因组和转录组学数据,并提供了多种基因组比较分析工具。自正式上线运行以来,MolluscDB已吸引了来自70多个国家的近15000次访问,成为全球范围软体动物研究的重要组学资源中心。

图1 MolluscDB数据库国际影响力(a: 访问国家;b: 引文领域;c: 数据库排名;d: 国际M10K计划启动)
近年来,高精度、多维度的功能基因组学数据呈爆发式增长,推动软体动物进入系统生物学时代,为软体动物科学研究带来新的发展机遇。然而,如何整合具有“复杂、高维、海量”特征的多组学资源,构建适用于软体动物生物学特性的定制分析平台,仍是国际软体动物研究领域共同面临的重要挑战。为了应对这一挑战,本研究团队将原有MolluscDB升级为MolluscDB 2.0,系统梳理整合软体动物复杂高维组学数据资源,致力开发丰富的可定制的系统生物学分析工具(包含近期开发的PanSyn工具包,Nature Protocols 2024),打造迄今最为系统全面的软体动物功能和进化基因组学综合分析平台。
MolluscDB 2.0收集并整合了近4200份多组学数据资源,实现主流组学维度的全覆盖,如高质量基因组、bulk转录组、单细胞转录组、蛋白质组、表观遗传组、微生物宏基因组等。软体动物多组学资源来自1450个物种,涵盖了软体动物门全部8个纲和76个目中的92%,地理分布覆盖从陆地、淡水、近海到深海,囊括了已公开的绝大部分软体动物的多组学资源。MolluscDB 2.0极大提升了原有14种基础分析模块,包括基因组组装信息、系统演化关系、古老化石记录、基因序列及结构、基因功能注释、发育时期/成体组织表达谱、基因家族、转录因子和转座子等。此外,针对软体动物的生物学和进化特性,MolluscDB还提供了多达20种满足特定研究需要的定制分析模块,包括泛进化综合分析模块、进化发育(evo-devo)综合分析模块和功能基因组综合分析模块(涵盖单细胞组学、蛋白组、表观修饰组、宏基因组)等。最终,通过将多维组学信息集成到开发定制的基因组浏览器中,实现了复杂多组学信息的便捷可视化和整合分析。
MolluscDB 2.0为软体动物研究领域提供一个物种覆盖度最广、组学资源最丰富、分析功能最全面的开放获取数据库平台,实现对复杂海量多组学资源的系统整合和深度分析,助力更全面地揭示软体动物的生物学奥秘和演化历程,推动认知海洋生物独特生命过程演变规律,也将为贝类重要基因资源发掘、遗传育种工作等提供有力支撑。
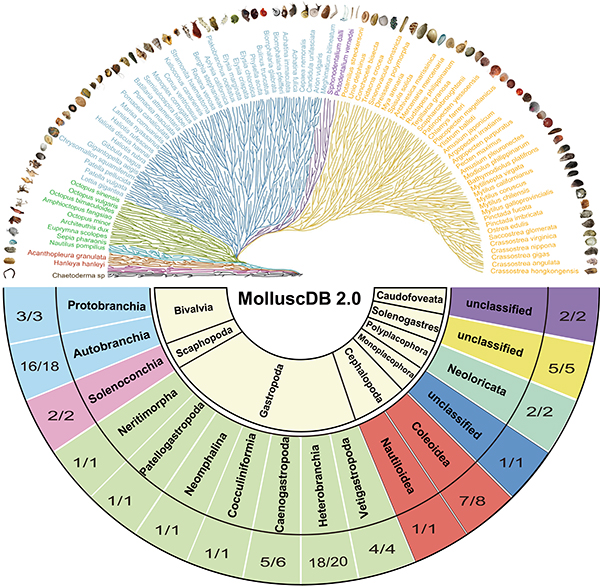
图2 MolluscDB 2.0数据库物种分类和覆盖情况总览
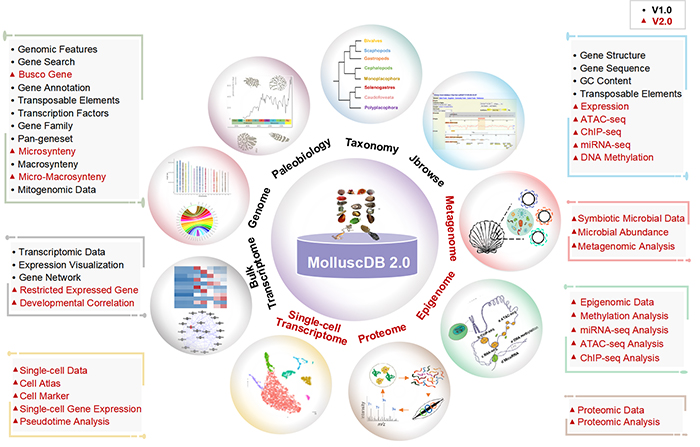
图3 MolluscDB 2.0数据库架构和多组学功能模块概览
海洋生物遗传学与育种教育部重点实验室、方宗熙海洋生物进化与发育研究中心的李语丽教授、王师教授、张玲玲教授为论文的共同通讯作者,刘福云博士、硕士生蔡柄丞、连姗姗副教授为论文共同第一作者。研究工作获得国家重点研发计划、国家自然科学基金、崂山实验室科技创新项目、山东省泰山学者等项目资助。该项工作同时获得了青岛海洋科学与技术试点国家实验室高性能科学计算与系统仿真平台、中国海洋大学高性能生物超算中心等单位的大力支持。
通讯员:王志刚
论文链接:https://doi.org/10.1093/nar/gkae1026
编辑:赵奚赟
责任编辑:刘莅




































